IDSC感染专栏54①|二代测序预测革兰阳性球菌耐药性有新发现
细菌耐药在全球范围内愈加广泛,革兰阳性球菌(G+)中的耐药现象日渐普遍[1,2]。早期、准确的病原学及敏感药物的检测成为临床迫切需求。传统的药敏试验耗时长、药物选择滞后,且针对培养受限的菌株无法进行检测[3]。
细菌耐药特征由抗生素耐药基因(ARGs)决定,检测ARGs的分子技术飞速发展[4]。宏基因组高通量测序技术(mNGS)可从临床标本中快速全面检测病原微生物的DNA和RNA序列,具有检测细菌ARGs的潜力[5-7]。笔者团队研究发现,mNGS技术检测G+球菌ARGs预测耐药表型有一定临床应用价值。
mNGS检出金葡菌三大耐药基因或可预测耐药性
研究回顾性分析2021.4-2022.4科室呼吸道感染患者249例。
纳入标准:(1)临床确诊存在肺部感染;(2)支气管镜下灌洗操作,支气管肺泡灌洗液(BALF)送检mNGS;(3)mNGS检测出G+球菌感染。共纳入249例患者,平均(54±15.54)岁,其中男性127例。mNGS共检测到G+球菌10种,包括肺炎链球菌(153/249,61.45%)、假肺炎链球菌(105/249,42.17%)、金黄色葡萄球菌(42/249,16.87%)、屎肠球菌(7/249,2.81%)、粪肠球菌(6/249,2.41%)(图1A)。共检出ARGs的G+球菌15株,其中,肺炎链球菌(3/15,20%)、金黄色葡萄球菌(9/15,60%)、屎肠球菌(1/15,6.67%)、粪肠球菌(2/15,13.33%)(图1B)。

图1 G+球菌和ARGs G+球菌检出率
Figure 1 Profile of gram positive bacteria and ARGsdetected in 249 BALF samples with mNGS
mNGS检出含ARGs金黄色葡萄球菌9例,其中传统微生物培养有3例阳性样本,在这3例含ARGs的金黄色葡萄球菌中,耐药基因blaZ、mecA和aac(6')-Ie-aph(2'')-Ia预测的耐药表型与传统药敏培养结果一致性较高,四环素耐药基因tet(L)一致性较差(表1)。
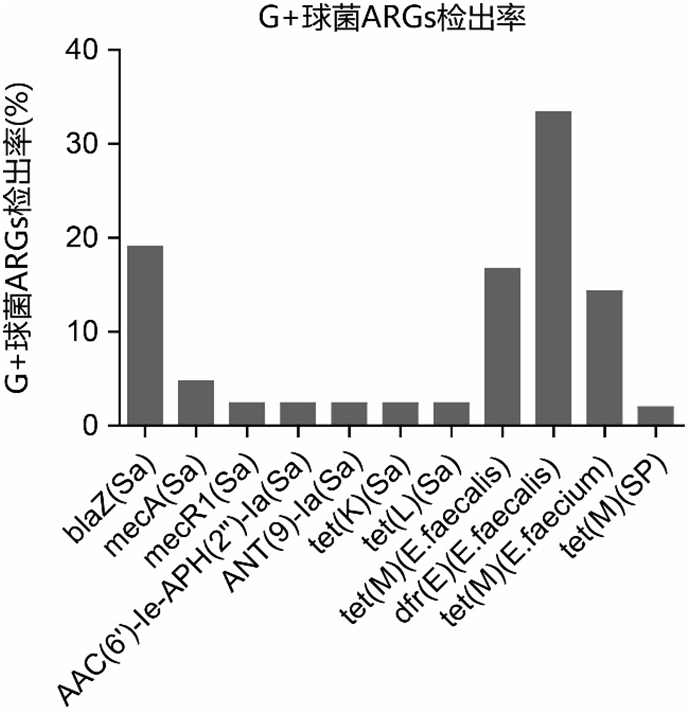
图2 G+球菌ARGs检出率
Figure 2 ARGs in gram positive bacteria identified by mNGS in clinical samples
Sa: streptococcus pneumoniae,金黄色葡萄球菌;E.faecalis: Enterococcus faecalis,粪肠球菌;E.faecium: Enterococcus faecium,屎肠球菌;SP: streptococcus pneumoniae,肺炎链球菌
表1 mNGS检出ARGs的金黄色葡萄球菌的耐药基因型和耐药表型
Table 1 Genotype and phenotype of staphylococcus aureus with ARGs detected by mNGS

mNGS深入分析和精准定位病原体是未来方向
随着耐药机制的深入研究,检测耐药基因的分子检测手段也在迅速发展。mNGS高通量测序技术可一次性、无偏差的对样本中整个微生物群的基因进行测序和分析,不仅可迅速、精准识别出致病微生物,还可全面、快速的鉴定细菌耐药基因。临床mNGS对致病菌耐药性质的判断仍处于摸索阶段。有研究借助mNGS新发现了9个通过降低酶活性导致四环素耐药的基因,进而揭示了四环素耐药新机制[8]。Rounak团队采用mNGS直接检测肺囊性纤维化患者痰液标本,发现mNGS不仅可以提供完整的微生物学信息,同时还能给予相对准确的耐药信息[9]。国内的一项多中心研究通过分析295份临床痰或血标本微生物培养及mNGS结果,发现mNGS检测出的抗菌药物耐药基因型与抗菌药物耐药表型间存在密切相关性[10]。这些研究提示mNGS对耐药基因检测具有临床应用潜力。
抗菌药物穿透力下降或外排增加、靶位点改变和水解酶使抗菌药物失活是细菌对抗菌药物耐药的三种主要机制,各种机制均存在与其相关的基因改变。金黄色葡萄球菌最常见耐药机制有两种,一是由blaZ基因编码产生β-内酰胺酶,对青霉素类耐药;二是mecA编码青霉素结合蛋白PBP2a,对甲氧西林耐药,mecA转录由mecR1/mecI调控。已有研究发现mecA耐药基因与耐甲氧西林金黄色葡萄球菌(MRSA)耐药表型有较高的一致性[11,12, 13]。本研究发现青霉素耐药基因BlaZ、甲氧西林耐药基因mecA、氨基糖苷类抗生素耐药基因aac(6')-Ie-aph(2'')-Ia可较好的预测耐药表型,四环素耐药基因tet预测耐药表型一致性较差。但如何正确解读mNGS耐药基因结果还需要更大样本量和更深入的研究。
mNGS检测耐药基因目前仍存在一些问题。首先,mNGS难以获取耐药基因全长序列信息,故对耐药基因亚型的深入分析受限;其次,病原体的一些共有基因的序列具有较高相似性,mNGS检测出的耐药基因可能无法精确定位至相应病原体;并且耐药基因数据库某些注释不准确,易出现基因-表型不完全匹配现象[14]。随着临床对测序技术深入研究和评价,mNGS指导下的精准抗感染治疗能够在感染性疾病诊治中发挥更大的作用。
参考文献
[1] Aslam B, Khurshid M, Arshad MI, Muzammil S, Rasool M, Yasmeen N, et al. Antibiotic Resistance: One Health One World Outlook. Front Cell Infect Microbiol. 2021;11:771510.
[2] Witte W. Antibiotic resistance in gram-positive bacteria: epidemiological aspects. Journal of Antimicrobial Chemotherapy. 1999;44(suppl_1):1-9.
[3] Didelot X, Bowden R, Wilson DJ, Peto TE, Crook DW. Transforming clinical microbiology with bacterial genome sequencing. Nature Reviews Genetics. 2012;13(9):601-12.
[4] Zankari E, Hasman H, Kaas RS, Seyfarth AM, Agersø Y, Lund O, et al. Genotyping using whole-genome sequencing is a realistic alternative to surveillance based on phenotypic antimicrobial susceptibility testing. Journal of antimicrobial chemotherapy. 2013;68(4):771-7.
[5] Yuan J, Li W, Qiu E, Han S, Li Z. Metagenomic NGS optimizes the use of antibiotics in appendicitis patients: bacterial culture is not suitable as the only guidance. American journal of Translational research. 2021;13(4):3010.
[6] Yee R, Breitwieser FP, Hao S, Opene BN, Workman RE, Tamma PD, et al. Metagenomic next-generation sequencing of rectal swabs for the surveillance of antimicrobial-resistant organisms on the Illumina Miseq and Oxford MinION platforms. European Journal of Clinical Microbiology & Infectious Diseases. 2021;40(1):95-102.
[7] Sukhum KV, Diorio‐Toth L, Dantas G. Genomic and metagenomic approaches for predictive surveillance of emerging pathogens and antibiotic resistance. Clinical Pharmacology & Therapeutics. 2019;106(3):512-24.
[8] Forsberg KJ, Patel S, Wencewicz TA, Dantas G. The tetracycline destructases: a novel family of tetracycline-inactivating enzymes. Chemistry & biology. 2015;22(7):888-97.
[9] Feigelman R, Kahlert CR, Baty F, Rassouli F, Kleiner RL, Kohler P, et al. Sputum DNA sequencing in cystic fibrosis: non-invasive access to the lung microbiome and to pathogen details. Microbiome. 2017;5(1):1-14.
[10] Chao L, Li J, Zhang Yn, Pu H, Yan X. Application of next generation sequencing-based rapid detection platform for microbiological diagnosis and drug resistance prediction in acute lower respiratory infection. Annals of Translational Medicine. 2020;8(24).
[11] Strauss C, Endimiani A, Perreten V. A novel universal DNA labeling and amplification system for rapid microarray-based detection of 117 antibiotic resistance genes in Gram-positive bacteria. Journal of microbiological methods. 2015;108:25-30.
[12] Pasanen T, Korkeila M, Mero S, Tarkka E, Piiparinen H, VUOPIO‐VARKILA J, et al. A selective broth enrichment combined with real‐time nuc‐mecA‐PCR in the exclusion of MRSA. Apmis. 2010;118(1):74-80.
[13] Shahmohammadi MR, Nahaei MR, Akbarzadeh A, Milani M. Clinical test to detect mec A and antibiotic resistance in Staphylococcus aureus, based on novel biotechnological methods. Artificial cells, nanomedicine, and biotechnology. 2016;44(6):1464-8.
[14] 梁文炎, 施毅, 孙文逵. 宏基因组高通量测序技术在革兰阴性菌耐药表型检测中的研究进展. 中华结核和呼吸杂志. 2022;45(02):209-13.
发表评论
最新评论

-
new教师节特辑 | 北京医院刘明主任寄语:“敬畏生命、敬畏专业、敬畏未知”
2025-09-10 -
04-292025
人工智能诊疗将走向前台——医生做好准备了吗
-
new肝癌免疫治疗的“路障”:胆汁酸 | 科普时光
2025-03-07 -
02-172025
慢病管理药先行 临床药师如何打通最后一公里
-

双镜合壁 助力消化疾病微创精准治疗
2024-12-24 -

朱玉龙:严重肺间质纤维化患者肺部结节经皮穿刺活检的注意事项
2024-11-21 -

IDSC感染专栏54③|重症感染患者弥散性血管内凝血早期诊断:中国诊断积分系统CDSS显优势
2022-06-28 -
IDSC感染专栏54②|夏秋季的烦恼,这种介于细菌与病毒间的病原在我国有抬头之势
2022-06-28 -
IDSC感染专栏54①|二代测序预测革兰阳性球菌耐药性有新发现
2022-06-28
-

何为巴林特小组?一文读懂
2017-02-23 -

IDSC感染专栏54③|重症感染患者弥散性血管内凝血早期诊断:中国诊断积分系统CDSS显优势
2022-06-28 -
益生菌预防艰难梭菌感染真的有效吗?
2021-09-07 -
孩子不是大人“缩微版” 小儿妇科聚焦未成年女性健康
2023-05-30 -

倪晓光:用好手中喉镜 提升鼻咽喉早癌检出率
2023-03-07 -
赵进喜教授:发挥中医药防治糖尿病肾脏病的特有优势
2023-06-09 -

心血管代谢医学|将临床研究融入社区日常临床诊疗以提高医学服务水平和质量
2021-11-05






